Entwicklung einer Plattform zur 3D-Visualisierung und -Segmentierung medizinischer Daten
Karl-Ingo Friese, Leibniz Universität Hannover
Visualisierung wird in der Wissenschaft in nahezu jeder Disziplin als Werkzeug für die Datenanalyse verwendet. Eine Tabelle führt präzise jedes Detail auf, die Tendenz wird jedoch in der Darstellung durch einen Graphen leichter erkennbar. Bei mehrdimensionale Daten führt oft erst ein Multiplotverfahren zum erhofften Erkenntnisgewinn. Die zu visualisierenden Daten sind dabei oft abstrakt. So wird bspw. die Druck- oder Temperaturverteilung in einem Werkstück in einer Falschfarbendarstellung visualisiert oder die regionale Preisentwicklung bei Lebensmitteln in einer Kombination aus Landkarte und Balkendiagramm skizziert.
Auch in der modernen Medizin fallen große Datenmengen an, insbesondere durch nichtinvasive 3D Bildgewinnungsmethoden (CT, MRI, Ultraschall, ...). Solche Aufnahmen werden zur Diagnose einer Krankheit oder einer Verletzung eingesetzt, zur konkreten Planung einer komplizierten Operation und zunehmend auch für die anschließenden Qualitätskontrolle. Für manche Aufgaben ist es wichtig, eine möglichst schnelle, für andere eine möglichst hochwertige Darstellung der gewonnenen Daten zu erzeugen. Besonders für die 3D-Segmentierung ist eine angemessene, intuitiv verständliche, exakte und vor allem interaktive Visualisierung der vorhandenen Volumendaten wichtig.
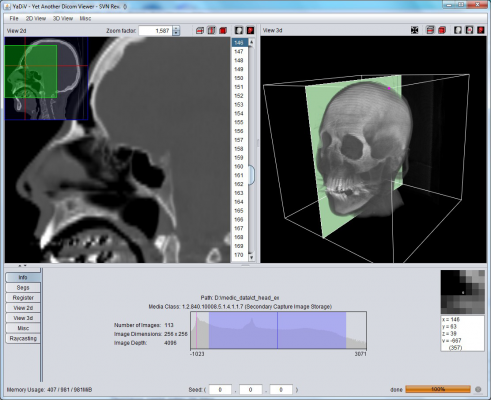
Ziel dieser Arbeit ist es, eine Visualisierungs- und Segmentierungsplattform für medizinische 3D Daten zu entwickeln. Dabei richtet sich das Programm vor allem an Wissenschaftler aus den Gebieten der medizinischen Visualisierung und Segmentierung, die neue Methoden - wie z.B. ein anspruchsvolles Segmentierungsverfahren oder die Erprobung neuer, haptischer Eingabemethoden - entwickeln wollen. Das Resultat dieser Arbeit ist das Programmpaket ”YaDiV”, das in Kürze als Open-Source Version der Wissenschaftsgemeinde zur Verfügung stehen wird.
Neben der leichten Erweiterbarkeit um neue Module, war Plattformunabhängigkeit eine der Hauptanforderungen, um möglichst vielen Wissenschaftlern die Möglichkeit zu geben, YaDiV als Grundlage für Ihre Arbeit zu nutzen. Ein weiteres Kriterium war konsequente Multi-Thread Unterstützung, um die zum Teil sehr aufwendigen Methoden (z.B. im Bereich Direct Volume Rendering oder Moving-Contour Segmentierung) möglichst optimal an die sich immer weiter verbreitenden Mehrprozessor- (oder zumindest Mehrkern-)Systeme anpassen zu können. Die zu Beginn der Entwicklung getroffene Entscheidung, Java als Programmiersprache zu verwenden, wurde besonders durch diese Anforderungen bestärkt.