Visualisierung
Unter dem Begriff "Visualisierung" versteht man Verfahren, die abstrakte Daten oder Zusammenhänge in eine graphische, visuell erfassbare Form bringen. Unser Forschungsschwerpunkt liegt hier auf der 3D-Visualsierung, d.h. der Darstellung von dreidimensionalen Objekten. Anwendungen hierfür sind u.a.
- Konstruktionsaufgaben in der Architektur oder im Maschinenbau,
- die Darstellung dreidimensionaler Daten in der Medizin, wie sie zum Beispiel durch einen Computertomographen erzeugt werden oder
- die Visualisierung von Landschaftsdaten - als reine Bestandsaufnahme oder als Modell für eine geplante Maßnahme.
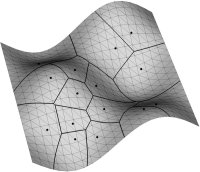
Schon kurz nach der Gründung unseres Lehrstuhls war 3D Visualisierung ein wichtiges Thema. So wurde 1994 das NURBS basierte Visualisierungsprogramm "Janus" ("Just Another NUrbs System") entwickelt, das neben NURBS Flächen auch die Darstellung von Bezierflächen (und -Kurven), Dreiecksflächen, Punktwolken oder Curve-On-Surface Modelle visualisieren kann. Auch für viele grundlegende geometrische Fragestellungen ist die Visualsierung ein wichtiges Hilfsmittel. Ob nun ein 3D Voronoi Diagram oder die mediale Achse berechnet werden soll, die erste Kontrolle ob ein Algorithmus das richtige Ergebnis liefert erfolgt meist graphisch.
3D Visualisierung im Bereich Medizin
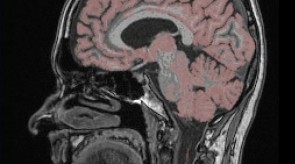
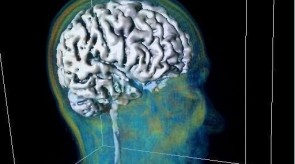
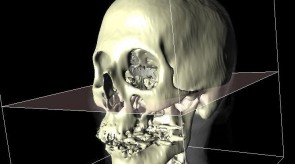
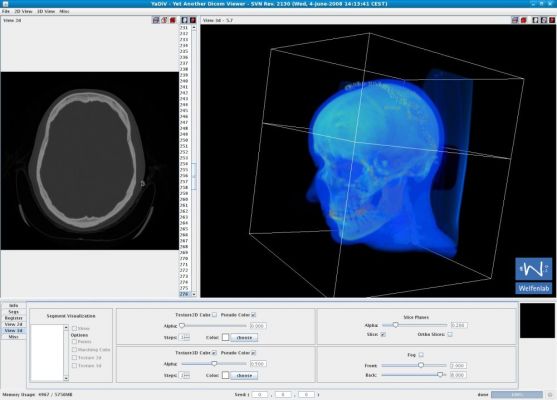
Ein besonders aktueller Schwerpunkt des Welfenlab ist die Visualisierung von Voxeldaten aus dem Bereich der Medizin. Unter einem Voxel kann man sich das dreidimensionale Gegenstück eines Pixels vorstellen. Während ein Pixel (bestehend aus einer x und y Koordinate zusammen mit einem Farbwert) einen Punkt auf einem Computermonitor beschreibt, besitzt ein Voxel zusätzlich eine z-Koordinate. Solche Voxeldaten werden u.a. durch verschiedene bildgebende Verfahren der Medizin gewonnen, z.B. durch Computertomographie (CT) oder Magnetresonanztomographie (MRT). Aus Sicht der Informatik stellt sich die Frage, wie solche Voxeldaten in einer für den Menschen verständlichen Art und Weise dargestellt werden können. Je nach Anwendung kommt es dabei entweder darauf an eine besonders schöne und leicht verständliche Visualisierung zu finden, oder eine möglichst schnelle, interaktive Darstellung bei der sich der Betrachter um das virtuelle Objekt herum oder sogar hindurch bewegen kann.
Zusammen mit dem Problem der Visualisierung tritt oft auch das Problem der Segmentierung auf. Unter diesem Begriff versteht man die Erzeugung von inhaltlich zusammenhängenden Regionen durch das Zusammenfassen von Pixeln oder Voxeln entsprechend einem bestimmten Homogenitätskriterium. Ein CT-Voxeldatensatz kann z.B. eine Aufnahme eines menschlichen Kopfes darstellen. Ein Teil dieser Aufnahme besteht aus Knochen, ein Teil des Knochens würde von einem Mediziner als "Unterkiefer" bezeichnet. Beides, sowohl "Knochen" als Oberbegriff als auch "Unterkiefer" als Teilmenge von "Knochen", bezeichnet man als Segment und die (manuelle oder automatische) Erzeugung eines solchen Segments als Segmentierung.
Beide Anwendungen, Visualisierung und Segmentierung werden am Welfenlab erforscht. Hierfür wurde mit dem Programm YaDiV ("Yet Another Dicom Viewer") eine eigene Software entwickelt, die DICOM Daten einlesen, anzeigen, segmentieren und registrieren kann. Die Evaluation der entwickelten Verfahren erfolgt durch Ärzte an der Medizinischen Hochschule Hannover MHH. Es ist geplant, YaDiV in den kommenden Wochen als OpenSource Software zu veröffentlichen.
Visualisierung mit Spiele-Engines
Ein weiteres Forschungsgebiet stellen Visualisierungen mit Spiele-Engines dar. Gerade in den letzten Jahren hat sich die Computerspieleindustrie zu einem grossen und wichtigen Markt entwickelt, in dem beeindruckende Summen für die Entwicklung neuer 3D-Spiele Engines zur Verfügung stehen. Im Gegensatz zu diesen Entwicklungskosten steht jedoch der relativ geringe Preis für das Endprodukt, da Computerspiele im Gegensatz zu professionellen Visualisierungsprogrammen für einen Massenmarkt konzipiert wurden. Die Idee, dieses Potential auch für andere, seriöse Anwendungen zu Nutzen liegt daher nahe ("serious gaming").